1. INTRODUCTION
The oak genus, a deciduous broad-leaved tree, provides important ecological services to the forest ecosystem (Plomion and Fievet, 2013) and supplies resources such as wood and biochemicals that are important to humans (Li et al., 2010; Chang et al., 2017). Out of the 1,372 species of them, Quercus mongolica has 308 trees as a dominant species that accounts for about 28.4%, and it has been reported that they are distributed throughout Korea (Lam et al., 2015; Lee et al., 2018) with the area of 229,821 ha (Korea Forest Service, 2018). The fruit of the Q. mongolica is edible, and its wood has been used economically and industrially as a raw material for building materials and equipment (Son et al., 2004). It contains biologically active substances with antibacterial, anti-inflammatory, and antioxidant properties, and is known as a species with high utilization values (Eo et al., 2019; Kong et al., 2001; Yin et al., 2019; Jung et al., 2017; Jung et al., 2017).
However, Q. mongolica is easily exposed to external wounds and microbial invasion because the cork later devolopment is relatively insufficient and the incidence of sprout is high. Therefore it has been reported as a species with a high motality rate by decay. (Lee et al., 2017; Jeon et al., 2020). According to previous reports, the decay rate of the 2,504 Q. mongolica harvested from nationwide in Korea was 66.1%, and most of them were in the stage of tissue death and discoloration. Also, it has been reported that the decay rate increases as the diameter and age of the tree increase (Kang et al., 2020). Q. mongolica with decay changes the color of inside to brown and dark brown color or formation of cavities resulting in the decrease in the quality of wood, so serious economic damage is caused. Despite these obstacles for economic uses, no clear cause of the decay has been elucidated. It is necessary to identify the cause of these phenomena and to find a method to control the decay for the increase in added value for Q. mongolica.
The deterioration of wood, including decay, is a phenomenon that physical and chemical properties decomposed or degenerated and is generally divided into two types: biological deterioration caused by microorganisms or insects and non-biological deterioration caused by other causes (Brischke et al., 2006; Brischke and Rapp, 2010; Teodorescu et al., 2017). Fungi, which are wood decay microorganism, are a major cause of biodegradation, which weakens the trunk, branches, and roots of wood (Brischke et al., 2006; White, 2004). The wood decay by fungi changes the color or strength of heartwood or sapwood, and such timbers have less value for use and cause economic loss (Schwarze, 2007). Generally, natural biological deterioration is a complex process involved with diverse environmental conditions and types of fungi. Fungi related to decay are confirmed differently the type of species or enzyme activities such as cellulase and laccase depending on the type of wood. Therefore, it is necessary to study decay fungi related to specific wood (Clausen, 1996; Hildén and Mäkelä, 2018; White, 2004).
Studies for fungi-related decay have been previously reported in Korea and Japan to prevent and diagnose the damage by wood-decaying fungi for larch which is a main economic species (Kim, 1996; Kim, 1997). Similar to the decay damages in larches, the decay of Q. mongolica is expected to be related to microorganisms. Therefore, in this study, the fungi from the decayed area of Q. mongolica were isolated and identified to confirm the fungi-related decay and discoloration. Also, we evaluated the identified cellulase and laccase enzyme activity and wood-decaying ability to confirm whether they can directly affect the decay of Q. mongolica.
2. MATERIALS and METHODS
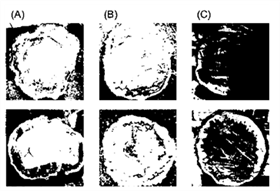
The Q. mongolica used in this study were collected from Hongcheon, Gangwon-do, Korea. The harvested timbers with decay or discoloration in the heartwood in the cross-section were selected (Fig. 1). They were transferred to the laboratory as slices and crushed into sawdust forms using a freeze-grinder (SPEXTM SamplePrep 6870 Freezer/MillTM, LLC, Metuchen, NJ, USA). During this process, samples for the isolation of fungi were prepared, and it took 1 week after harvesting.
Two independent experiments were performed to isolate fungi among microorganisms in the sawdust. As one experiment, 10 g of sawdust was put in the center of PD agar plate (DifcoTM potato dextrose agar, catalog number: 213400, Becton, Dickinson and Company, Franklin Lakes, NJ, USA), and the growth of microorganisms around it was monitored. As for the other experiment, 10 g of sawdust was vigorously mixed in 100 mL of 0.7% NaCl, and 10x serial dilutions were performed. 100 μL of each dilution was dispensed on PD agar plate and evenly spread using a glass spreader. PD agar plates prepared by these two methods were cultured at 25°C and a single fungus was separated depending on the growth rate.
Genomic DNA was extracted from the isolated and cultured fungi from the previously described method (Umesha et al., 2016). After transferring the fungi to a 1.5 mL Eppendorf tube, they were strongly ground with a sterilized stainless steel rod. 300 μL of extraction buffer (200 mM Tris-HCl (pH 8.0), 250 mM NaCl, 25 mM EDTA (pH 8.0), 0.5% SDS) was added to them and mixed by vigorous vortex. Samples were centrifuged at 4°C, at 13,000 rpm for 15 minutes using 5417R Refrigerated Centrifuge (Product number: 5407 14618, Eppendorf Scientific, Germany). 250 μL of the supernatants were transferred to fresh Eppendorf tubes and 250 μL of phenol-chloroform-isoamyl alcohol (25:24:1 Phenol:Chloroform:Isoamyl alcohol, Catalog number: C-9017, Bioneer, Korea) was added. After gentle mixing by converting, they were centrifuged at 4°C and 13,000 rpm for 5 minutes. 500 μL of the supernatants and 500 μL of cold ethanol diluted with 95% (99.5%, Catalog number: 056-03341, Wako, Japan) were added and mixed gently. After centrifuging them at 4°C, at 13,000 rpm for 20 minutes, ethanol was removed. After 1 mL of cold 70% ethanol was added and slowly mixed, samples were centrifuged at 4°C and 13,000 rpm for 10 minutes. After ethanol was completely removed, samples were dried with a cap left opened at room temperature for 1 hour. Samples were re-suspended with 30 μL of sterilized distilled water containing 10 μg/mL RNase (RNase A Powder for K-3030, Catalog number: KB-0101, Bioneer, Korea) and reacted at 37°C for 30 minutes to obtain genomic DNA.
To amplify Internal Transcribed Spacer (ITS) region from extracted genomic DNA, AccuPower® PCR PreMix (Catalog number: K-2016, Bioneer, Korea) was used, and reaction conditions were followed by the manual. Two primers (ITS1: 5’-TCCGTAGGTGAACCTGCGG-3’, and ITS4: 5’-TCCTCCGCTTATTGATATGC-3’) were used. The amplified PCR products were purified using AccuPrep® PCR/Gel DNA Purification Kit (Catalog number: K-3037, Bioneer, Korea) according to the manual. The sequencing of purified DNA was performed at National Instrumentation Center for Environmental Management (NICEM, Seoul, Korea) using ABI 3730x1 DNA analyzer (Catalog number: A41046, Thermo Fisher Scientific Korea Ltd., Korea). Homology analyses were performed on the produced sequence using nucleotide BLAST search at National Center for Biotechnology Information (NCBI).
Among homology search results of five fungi through nucleotide BLAST using nucleotide sequence obtained from PCR with their ITS regions as primers, the one with the highest homology was selected for each. Multiple sequence alignment for the selected nucleotide sequences was performed using ClustalW of the MEGA4 program. Neighbor-Joining (NJ) method was used for phylogenetic tree analysis of aligned nucleotide sequences, and bootstrap (1,000 replications) was applied for the reliability of the results.
The pH and temperature-dependent growth of fungi was monitored in PD agar plates with 90 mm diameter. Fungi were inoculated at the center of each PD agar plate with controlled pH conditions for 4 pH values (4.5, 5.0, 5.5, and 6.0) and cultured at 3 temperature conditions (25, 28, and 30°C) for each. All experimental results were calculated using IBM® SPSS software (Ver. 25.0; SPSS Inc., Chicago, Illinois, USA) for means and standard deviations. Results were analyzed by Duncan test method of One-way analysis at the 95% confidence level. The culture period of each fungi was determined as 80% of the plate area was covered by mycelium and the diameter of the ring at this time was measured to compare the growth rate of each fungus according to the culture conditions.
Formula: Growth rate (cm/day) = mycelium diameter (cm) / culture period (day)
Where, the diameter of a mycelium and culture period are based on when about 80% of the plate is filled with fungi. Also, the average mycelium diameter formed under all culture conditions is used.
Among the extracellular enzymes secreted by the fungus, the activities of cellulase and laccase were measured (Jeon and Ka, 2014). An inoculum of 6 mm diameter size was isolated and prepared from the strains cultured at 25°C on a PD agar plate. Carboxymethylcellulose sodium salt (CMC, Product number: C5678, Sigma, Korea) was selected as a substrate for the cellulase activity measurements. An inoculum with 6 mm diameter was inoculated on the center of each 50 mm diameter CMC agar (0.2% NaNO3, 0.1% K2HPO4, 0.05% MgSO4, 0.05% KCl, 0.2% carboxymethylcellulose sodium salt, 0.02% peptone, 1.5% agar, w/v) plate and cultured at 25°C in the darkness for 2 days. Then, 1-2 mL of Gram’s iodine solution (0.67% KI, 0.003% I2, w/v) was poured onto each plate and reacted at room temperature in the darkness for 2 hours. After 2 hours, the diameter of the zone showing cellulase activity was measured. 2,2’-azino-bis(3-ethylbenzthiazoline-6-sulfonic acid) diammonium salt (ABTS, Product number: A1888, Sigma, Korea) was used as a substrate for the laccase activity measurements. An inoculum with 6 mm diameter was inoculated on the center of each 90 mm diameter ABTS agar (0.2% K2HPO4, 0.2% KCI, 0.2% MgSO4·7H2O, 1.5% agar, pH 5.0, w/v) plate and cultured at 25°C in the darkness for 5 days. Five days later, the size of the turquoise blue ring formed around the inoculum was measured.
The wood-decaying ability by fungi on Q. mongolica was evaluated following KS F 2213 : 2004 standard. A testing strain for broad-leaved trees, Trametes versicolor was used as a control group (Won et al., 2018). About 250 g of quartz sand dried after washing with water was filled in a 500 mL glass bottle. 80 mL of culture medium (2.5% glucose, 1% malt extract, 0.5% peptone, 0.3% KH2PO4, 0.2% MgSO4·7H2O) was evenly absorbed and sealed, and then sterilized at 121°C for 30 minutes. Heartwood and sapwood of Q. mongolica were prepared in a size of 20 x 20 x 20 mm, respectively. The prepared samples were dried at 60°C for 48 hours, then allowed to cool down in a desiccator for 30 minutes (conditioning), and their weight (W1) values were measured. Five strains of fungi were cultured for 5 days in PD (DifcoTM potato dextrose broth, catalog number: 254920, DB, USA) broth at 25°C and 150 rpm conditions, and 3 mL of culture were evenly applied to the surface of the quartz sand. Three prepared samples were put in each bottle, and then decayed at 25°C and a humidity of 70% or more, for about 40 days. The fungi adhering to the sample surface were removed thoroughly with a soft brush in flowing water and air-dried for about 20 hours. Then, they were dried at 60°C temperature condition for 48 hours, and then allowed to cool down in a desiccator for 30 minutes (conditioning), and their weight (W2) values were measured. The weight reduction rate was calculated by the formula below, and the decay ability of five fungi strains was evaluated.
Formula: Weight reduction rate (%) = (W1 – W2)/W1 x 100
W1: weight of the sample before fungal inoculation
W2: weight of the sample after fungal inoculation
3. RESULTS and DISCUSSION
The nucleotide sequences were obtained by PCR amplification of the ITS1 and ITS4 regions of genomic DNA extracted from the fungi. Obtained nucleotide sequences were compared to pre-existing databases using NCBI, nucleotide BLAST search. As a result (Table 1) of analyzing the nucleotide sequence homology of the fungi isolated from the decaying wood shown in Fig. 1(A), Fungal sp. was ranked at the top, followed by Umbelopsis isabellina. The nucleotide sequence of fungi isolated from the decaying wood illustrated in Fig. 1(B) confirmed them to be fungi belonging to Ophiostoma species and Ophiostoma piceae accounted for 100% in query coverage and identities. Three morphologically distinct three fungi were isolated from the decaying wood shown in Fig. 1(C). The homology comparison from one of them showed that all of the top five were identified as Aureobasidium melanogenum, and query coverage and identities were 99%. Another was confirmed to be Mucor circinelloides, and query coverage and identities were 99%. The third also showed that all of the top 5 were identified as Cunninghamella elegans, and the top result showed 99% in query coverage and 100% identities. Based on these results, the isolated fungi were suggested to be O. piceae and A. melanogenum which belong to Ascomycota phylum, and M. circinelloides, U. isabellina and C. elegans, three species that belong to Mucoromycota phylum.
| Origin | Description | Max / Total score | Query coverage / Identities (%) |
E-value | Accession |
|---|---|---|---|---|---|
| Fig. 1(A) | Fungal sp. 3.12.4D | 1109 / 1109 | 99 / 99 | 0.0 | KM068430.1 |
| Umbelopsis isabellina | 1083 / 1083 | 99 / 98 | 0.0 | KC489502.1 | |
| Umbelopsis isabellina | 1083 / 1083 | 99 / 98 | 0.0 | AJ876493.1 | |
| Umbelopsis isabellina | 1077 / 1077 | 99 / 98 | 0.0 | KJ028792.1 | |
| Fungal endophyte sp. P1406O | 1077 / 1077 | 98 / 99 | 0.0 | EU977237.1 | |
| Fig. 1(B) | Ophiostoma piceae | 1188 / 1188 | 100 / 100 | 0.0 | KF531618.1 |
| Ophiostoma quercus | 1179 / 1179 | 99 / 100 | 0.0 | KM100571.1 | |
| Ophiostoma piliferum | 1175 / 1175 | 100 / 99 | 0.0 | KU756609.1 | |
| Ophiostoma querci | 1175 / 1175 | 100 / 99 | 0.0 | AF493243.1 | |
| Ophiostoma quercus | 1173 / 1173 | 99 / 99 | 0.0 | MH055670.1 | |
| Fig. 1(C) | Aureobasidium melanogenum | 1068 / 1068 | 99 / 99 | 0.0 | KY659501.1 |
| Aureobasidium melanogenum | 1068 / 1068 | 99 / 99 | 0.0 | KY294711.1 | |
| Aureobasidium melanogenum | 1068 / 1068 | 99 / 99 | 0.0 | KY294710.1 | |
| Aureobasidium melanogenum | 1068 / 1068 | 99 / 99 | 0.0 | KY294709.1 | |
| Aureobasidium melanogenum | 1068 / 1068 | 99 / 99 | 0.0 | KY294708.1 | |
| Fig. 1(C) | Mucor circinelloides | 1177 / 1177 | 99 / 99 | 0.0 | JN561250.1 |
| Mucor circinelloides | 1170 / 1170 | 99 / 99 | 0.0 | DQ118987.1 | |
| Mucor circinelloides | 1168 / 1168 | 99 / 99 | 0.0 | KP132465.1 | |
| Mucor circinelloides | 1166 / 1166 | 99 / 99 | 0.0 | KY933391.1 | |
| Mucor circinelloides | 1164 / 1164 | 99 / 99 | 0.0 | GU966516.1 | |
| Fig. 1(C) | Cunninghamella elegans | 1245 / 1245 | 99 / 100 | 0.0 | JN205887.1 |
| Cunninghamella elegans | 1240 / 1240 | 99 / 100 | 0.0 | JN205882.1 | |
| Cunninghamella elegans | 1240 / 1240 | 99 / 99 | 0.0 | FJ792589.1 | |
| Cunninghamella elegans | 1234 / 1234 | 99 / 99 | 0.0 | JF439470.1 | |
| Cunninghamella elegans | 1232 / 1232 | 99 / 99 | 0.0 | JN205884.1 |
Phylogenetic tree analysis was performed by selecting one each with the highest max/total score and query coverage/identities from the homology search results using the nucleotide sequences of the five fungi obtained (Fig. 2). As a result, O. piceae (KF531618.1) and A. melanogenum (KY659501.1) were grouped into one group, M. circinelloides (JN561250.1), U. isabellina (KC489502.1), and C. elegans (JN205887.1) formed another group. These two groups were separated in the early stage, and it was confirmed that the similarity between these two lines was quite distant. In one group, M. circinelloides and U. isabellina were bound to the same branch, but C. elegans initially formed different branches, meaning that C. elegans had a distance in lineage from the other two species. On the

other hand, A. melanogenum and O. piceae showed a value of 100, which was considered to have high similarity in their nucleotide sequences.
Five morphological distinct fungi were isolated and cultured from deteriorated Q. mongolica at 25°C temperature condition using PD agar as a nutrient source (Fig. 3). After observation of mycelia from 5 fungi, the surface texture of M. circinelloides and C. elegans was downy surface texture (Fig. 3(A) and 3(B)). Also, in the case of U. isabellina (Fig. 3(C)), the surface of the formed mycelium showed a rough texture. On the other hand, the mycelium of O. piceae and A. melanogenum has a relatively flat surface, smooth texture, and glossy (Fig. 3(D) and 3(E)). Also, M. circinelloides, C. elegans and U. isabellina had unclear boundaries of their mycelia, but O. piceae formed dark circular bands like annual ring around the inoculum, as the mycelium spread. In addition, A. melanogenum showed bright color in early stage mycelium but showed a tendency to darken over time forming dark brown color around the inoculum. In summarizing the morphological characteristics, it was confirmed that M. circinellodes, C. elegans, and U. isabellina formed one group, and O. piceae and A. melanogenum formed the other group. This result of being divided into two groups supported well the result of phylogenetic tree analysis using nucleotide sequences (Fig. 2), which were classified into Mucoromycota phylum and Ascomycota phylum. If the characteristics of fungi are investigated through the unique morphological analysis, it is considered that it will be useful for the initial classification of strains before a genetic identification process.

In order to observe the growth characteristics of the five fungi strains isolated and identified from Q. mongolica depending on the culture conditions, fungi were inoculated into PD agar plates adjusted to various pH values (pH 4.5, pH 5.0, pH. 5.5, and pH 6.0) and incubated at temperature conditions of 25°C, 28°C, and 30°C. The inoculated fungi were incubated until the surface of a plate was more than 80% covered, and the incubation period was varied according to the growth rate of each strain. Also, the diameter of their mycelia was measured to calculate the growth rate according to the cultivation period to compare the growth rate among fungi (Table 2).
After checking the growth rate for each species, it took 3 days until 80% of the plate surface was covered with mycelium by M. circinellodes and C. elegans, and the growth rate were confirmed to be 2.4 cm/day and 2.1 cm/day, respectively. Also, U. isabellina was required 9 days of the culture period, and its growth rate was confirmed to be 0.8 cm/day. O. piceae and A. melanogenum needed 17 days and 14 days to form sufficient mycelia, respectively, and the determined growth rate was confirmed to be 0.4 cm/day and 0.5 cm/day. From these results, the growth rates of M. circinelloides, C. elegans, and U. isabellina belonging to Mucoromycota phylum isolated and identified through this study were found to be 2 – 6 times faster than those of O. piceae and A. melanogenum belonging to Ascomycota phylum.
Growth changes depending on pH and temperature conditions were examined for each species, and basic physiological characteristics of each strain were investigated by observing a significant difference in growth with respect to the size of the mycelium measured under each condition. As a result, no significant pH/temperature dependence was observed from M. circinelloides and C. elegans. In contrast, U. isabellina formed a wider mycelium cultured at pH 6.0 than pH 4.5, at both 25°C and 30°C culture temperatures (p < 0.05). Based on these results, it is suggested that pH is a more important factor for the growth of U. isabellina than temperature. In the case of O. piceae, a wider mycelium was formed when cultured at pH 5.5 and 25°C than pH 5.5 and 30°C within the same period (p < 0.05). It is considered that relatively low temperature is suitable for the formation of O. piceae mycelium. The growth of A. melanogenum generally formed a wider mycelium when cultured at 25°C than 30°C (p < 0.05). From these results, it is suggested that relatively low temperatures are suitable for the growth of A. melanogenum.
Despite some differences in the culture period, all strains showed active growth enough to cover all the plates surface regardless of the culture conditions. This phenomenon is suggested that glucose, which is the main constituent of PD agar used for culture in this study, is the preferred energy source by most microorganisms, and therefore optimal culture conditions have not been derived. It is considered that further investigations will be necessary to observe the nutrient sources favored by the five fungi and the morphological characteristics expressed at that moment, by varying the medium conditions depending on carbon and nitrogen sources.
Woods have complex layered structures composed of polymer compounds of cellulose, hemicellulose, and lignin. Wood decaying fungi and other wood-harming microorganisms must have a system which is capable of decomposing these chemical structures to effectively decompose woods and obtain nutrients (Bidlack et al., 1992; Brändström, 2001; Yoon et al., 2018). Since the five fungi strains obtained in this study were isolated from deteriorated Q. mongolica, with decay and discoloration, it was expected that they would be able to decompose wood biomass. So, the presence or absence of enzymes that degrade cellulose and lignin were confirmed by using CMC and ABTS as substrates, respectively.
CMC is one of the cellulose derivatives (Hankin and Anagnostakis, 1977), and its β-1,4-glucosidic bond is hydrolyzed by cellulase to form glucose (Percival Zhang et al., 2006; Yoon et al., 2019). By inoculating 5 types of fungi on a 5.0 cm diameter agar plate containing CMC and incubating at 25°C, a ring was formed by cellulase activity around all inocula (Fig. 4(A)). Especially, M. circinelloides and C. elegans formed the biggest rings with the size of 4.3 and 4.0 cm, respectively. In contrast, U. isabellina, O. piceae, and A. melanogenum formed relatively small rings, 2.0, 1.9, and 1.8 cm, respectively. M. circinelloides and C. elegans, which formed big rings, belong to Mucoromycota phylum, and O. piceae and A. melanogenum, which formed relatively small rings, are classified as Ascomycota phylum. Laccase is an extracellular enzyme produced by most fungi and has been reported to break down lignin in wood (Baldrian, 2006; Giardina et al., 2010). By confirming the secretion of laccase, a lignin-degrading (ligninolytic) enzyme, with ABTS assay, the possibility of lignin decomposition capability of five fungi strains was confirmed. After inoculating strains on the agar plate containing ABTS and incubating for 5 days, the secretion of laccase was confirmed by the presence or absence of a turquoise ring (Fig. 4(B)). As a results, turquoise rings of 3.2 cm and 2.1 cm were formed around the inoculum of O. piceae and A. melanogenum, respectively. On the other hand, M. circienlloides, C. elegans, and U. isabellina did not form a ring induced by laccase decomposition. Interestingly, the two fungi strains, O. piceae and A. melanogenum, which their laccase secretion was confirmed by the formation of turquoise rings, belong to Ascomycota phylum, and the other three strains that do not secrete laccase belong to Mucoromycota phylum.
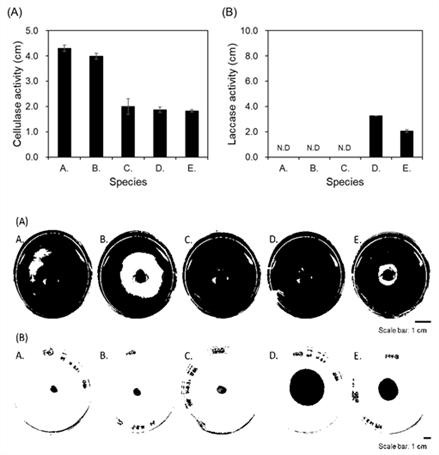
M. circinelloides, which is commonly found in soil, plants, and decayed fruits (Carvalho et al., 2015; Lee et al., 2014), has the nucleotide sequence of β-glucosidase, a type of cellulase, and it has been reported to show enzymatic activities (Huang et al., 2014). The M. circinelloides strain isolated and identified in this study also exhibited cellulase activity, and it is presumed to be a fungus involved with the deterioration of Q. mongolica. C. elegans has lignin peroxidase enzyme, and it has been reported to show the maximum enzyme activity at 30°C temperature and pH 6.0 conditions (Roushdy et al., 2011). However, the C. elegans strain obtained in this study did not show ligninolytic activity, and it might be explained to be caused by the difference in conditions for measuring the enzyme activity. Thus, it was considered that it is necessary to perform different temperature and pH conditions in the screening process to evaluate the presence or absence of enzyme activity as well. O. piceae, in which cellulase and laccase activities were observed, is a soft-rot fungus and has been reported to cause discoloration as well as decay of woods (Abraham et al., 2011). On the other hand, U. isabellina and A. melanogenum have not been reported on wood-degrading enzymes. However, in this study, cellulase activity of U. isabellina and cellulase and laccase activity of A. melanogenum were observed, so they would be related to the wood decay. Based on these results, among 5 fungi strains isolated from Q. mongolica in this study, it is expected that O. piceae and A. melanogenum, which belong to Ascomycota phylum, secrete both cellulase and laccase enzymes, involving with degrading cellulose and lignin of Q. mongolica.
In addition, it is considered that M. circinellodes, C. elegans, and U. isabellina, belonging to the Mucoro-mycota phylum, secrete cellulase and affect cellulose decomposition in Q. mongolica.
All of the five fungi isolated from the deteriorated Q. mongolica possessed cellulase activity. O. piceae and A. melanogenum among them showed laccase activity as well as cellulase activity. Based on these results, the 5 fungi identified in this study are expected to be related to the deterioration of Q. mongolica, so their decaying ability was experimentally examined on Q. mongolica. Because fungi with enzyme activity decompose the constituents of wood and use them as a nutrient source, the weight of woods decayed by them should be reduced. Therefore, the weight reduction rates of five strains of fungi were measured for heartwood and sapwood of Q. mongolica, and used as a criterion for evaluating the wood-decaying ability (Fig. 5). Results showed that the weight reduction rate of Q. mongolica heartwood by O. piceae was about 3.9% and sapwood was about 3.0%. On the other hand, the rest of the strains had insignificant effects on weight reduction rate which were showing less than 1% reduction for both heartwood and sapwood. When comparing the weight reduction rate by each fungus for the heartwood and sapwood, O. piceae and A. melanogenum, belonging to Ascomycota phylum, showed 6.9% and 1.4%, respectively, which were relatively higher than strains belonging Mucoromycota phylum: M. circinelloides (0.7%), C. elegans (1.3%), and U. isabellina (1.0%). Fungi belonging to Ascomycota phylum are also called soft-rot fungi, and they are found in both softwoods and hardwoods, but found more in hardwoods and are known to cause more damages. It has been reported that wood contaminated with soft-rot fungi undergo damages including softening surface and discoloration to dark brown (Nilsson and Daniel, 1989; Song et al., 2010).
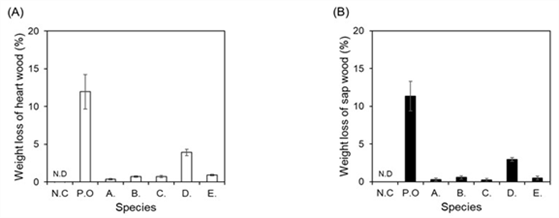
As previously described, all 5 fungi strains showed cellulase activity confirmed by CMC assay and ABTS assay, and two fungi belonging to Ascomycota phylum were found to possess laccase activity as well as cellulase activity. Lignin surrounds cellulose, and hemicellulose acts as a bridge between the two bonds (Carrier et al., 2017; Zhang et al., 2015). This tightly bonded lignin maintains the shape of a wood or protects cellulose from external invasion (Xie et al., 2018). So, the decomposition of lignin by the laccase enzyme activity helps the decaying microorganisms with enzymatic activity including cellulase to penetrate into the wood, suggesting the possibility that the decay of woods can proceed more effectively. Therefore, it is considered that the decomposition of lignin by laccase activity as well as cellulase activity acts as an important factor in the decaying of Q. mongolica, and it is suggested that O. piceae and A. melanogenum, belonging to Ascomycota phylum, which possess both enzyme activities, play a more important role in it.
4. CONCLUSION
From this study, overall five fungi strains were isolated and identified from Q. mongolica with discoloration, decay, and deterioration damages. The identified fungi were confirmed to be three species that belong to Mucoromycota phylum including M. circinelloidesC. elegans and U. isabellina, and two species that belong to Ascomytoca phylum including O. piceae and A. melanogenum. By evaluating the enzyme activity that affects wood decay, cellulase activity involved in cellulose degradation was observed in all fungi. Also, unlike the other fungi, O. piceae and A. melanogenum showed cellulase activity as well as laccase activity that degrades lignin. The weight reduction effect was also observed by all five fungi in the evaluation of wood decaying performed on the heartwood and sapwood of Q. mongolica. Based on these results, it is considered that the five fungi isolated from the deterioration of Q. mongolica have enzymes that degrade cellulose and lignin, which are major components of wood, and thus can affect wood decaying. Therefore, the identified fungi are considered to be directly involved with the decay of Q. mongolica, but in order to control the decay, further studies on the pathways and mechanisms by which these fungi are infected in living Q. monoglica will be needed.